lundi, 16 janvier 2023
Journal de bord de notre cerveau à tous les niveaux : la relation circulaire entre les gènes et le métabolisme
Comme je l’évoquais dans mon billet de la semaine dernière, je compte alimenter encore dans les mois qui viennent mon « journal de bord » de l’écriture de mon livre qui entre dans sa phase de relecture finale. Et l’une des choses qu’on savait déjà depuis longtemps mais que j’ai dû m’avouer officiellement récemment, c’est que c’est beaucoup trop long ! Je ne vous dirai pas le nombre de page qu’il fait actuellement de peur de vous effrayer, et n’évoquerai que vaguement les plusieurs centaines de pages de notes avec lesquelles je commençais à écrire chaque chapitre (et il y en a 12, plus prologue et épilogue…), mais il eut tout un travail de synthèse à faire pour arriver à la version actuelle qui est, donc, encore trop longue. Que faire alors ? Enlever des phrases pas essentielles ici et là ? À ce rythme, je n’y arriverai jamais… Il me fallait enlever carrément quelques « gros morceaux » par chapitre pour espérer voir le nombre de page diminuer substantiellement, la force d’un bras humain pour tenir un livre ayant ses limites… Chaque chapitre comportant plusieurs encadrés venant donner certains compléments d’information, on a donc décidé d’en retirer certains et de les transformer en… billets de blogue ! (suite…)
Du simple au complexe | Pas de commentaires
lundi, 14 décembre 2020
Percée majeure dans notre compréhension du repliement des protéines
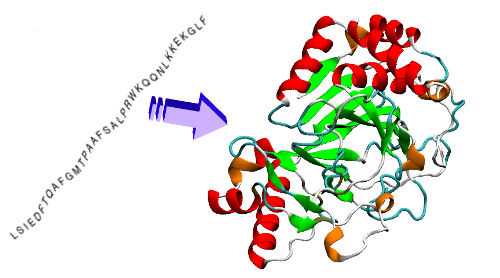 Le 30 novembre dernier, les résultats d’une compétition de prédiction de la structure des protéines appelée « CASP » (Critical assessment of structure prediction) ont été rendus publics. À chaque deux ans, une centaine d’équipe dans le monde tentent de découvrir à l’aide de logiciels la structure tridimensionnelle de certaines protéines que l’on a réussi à identifier par d’autres moyens expérimentaux. Depuis 2006, les taux de succès de ces logiciels oscillaient entre 30 et 40%. Puis, en 2018, le logiciel AlphaFold de la branche Deep Mind de Google, qui utilisent les réseaux de neurones artificiels (le fameux « deep learning »), a fait une entrée fracassante dans la partie avec un taux de succès de près de 60% ! C’est la même approche que Deep Mind avait déployé dans son logiciel AlphGo qui avait battu en 2016 le champion du monde du jeu de Go. Or les performances d’AlphaFold(2), la version 2020 du logiciel, lors de la compétition de cette année marquent, selon certains, un tournant majeur dans l’un des plus grands défis de la biologie puisque le logiciel a décrit la structure tridimensionnelle des protéines avec un taux de succès médian de 92% pour l’ensemble des protéines soumises et de 87% pour les protéines dont la structure était jugée comme étant particulièrement difficile à résoudre ! Est-ce le début d’une nouvelle ère ? C’est ce qu’affirment en tout cas certaines personnes qui travaillent dans le domaine. (suite…)
Le 30 novembre dernier, les résultats d’une compétition de prédiction de la structure des protéines appelée « CASP » (Critical assessment of structure prediction) ont été rendus publics. À chaque deux ans, une centaine d’équipe dans le monde tentent de découvrir à l’aide de logiciels la structure tridimensionnelle de certaines protéines que l’on a réussi à identifier par d’autres moyens expérimentaux. Depuis 2006, les taux de succès de ces logiciels oscillaient entre 30 et 40%. Puis, en 2018, le logiciel AlphaFold de la branche Deep Mind de Google, qui utilisent les réseaux de neurones artificiels (le fameux « deep learning »), a fait une entrée fracassante dans la partie avec un taux de succès de près de 60% ! C’est la même approche que Deep Mind avait déployé dans son logiciel AlphGo qui avait battu en 2016 le champion du monde du jeu de Go. Or les performances d’AlphaFold(2), la version 2020 du logiciel, lors de la compétition de cette année marquent, selon certains, un tournant majeur dans l’un des plus grands défis de la biologie puisque le logiciel a décrit la structure tridimensionnelle des protéines avec un taux de succès médian de 92% pour l’ensemble des protéines soumises et de 87% pour les protéines dont la structure était jugée comme étant particulièrement difficile à résoudre ! Est-ce le début d’une nouvelle ère ? C’est ce qu’affirment en tout cas certaines personnes qui travaillent dans le domaine. (suite…)
Du simple au complexe | Comments Closed
mardi, 14 avril 2015
De quoi sont faits nos chromosomes ?

C’est en faisant une recherche sur Internet avec l’expression « De quoi sommes-nous faits ? » que je suis tombé par hasard sur le remarquable site web suisse Chromosome Walk (1er lien ci-bas). La page « De quoi sommes-nous faits ? » de ce site permet, en déplaçant de gauche à droite un curseur au bas de la page, de passer de la cellule aux chromosomes, puis à l’ADN dont ils sont constitués, à la copie en l’ARN messager de cet ADN capable de sortir du noyau cellulaire pour rejoindre les ribosomes capables à leur tour de construire les protéines correspondantes, ouf !… (suite…)
Du simple au complexe, Le bricolage de l'évolution | Comments Closed