lundi, 14 décembre 2020
Percée majeure dans notre compréhension du repliement des protéines
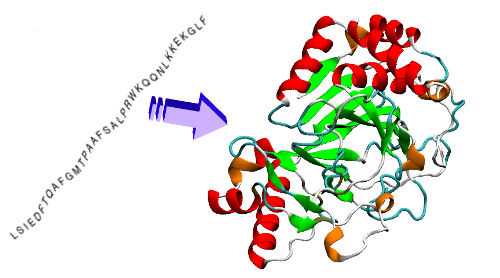 Le 30 novembre dernier, les résultats d’une compétition de prédiction de la structure des protéines appelée « CASP » (Critical assessment of structure prediction) ont été rendus publics. À chaque deux ans, une centaine d’équipe dans le monde tentent de découvrir à l’aide de logiciels la structure tridimensionnelle de certaines protéines que l’on a réussi à identifier par d’autres moyens expérimentaux. Depuis 2006, les taux de succès de ces logiciels oscillaient entre 30 et 40%. Puis, en 2018, le logiciel AlphaFold de la branche Deep Mind de Google, qui utilisent les réseaux de neurones artificiels (le fameux « deep learning »), a fait une entrée fracassante dans la partie avec un taux de succès de près de 60% ! C’est la même approche que Deep Mind avait déployé dans son logiciel AlphGo qui avait battu en 2016 le champion du monde du jeu de Go. Or les performances d’AlphaFold(2), la version 2020 du logiciel, lors de la compétition de cette année marquent, selon certains, un tournant majeur dans l’un des plus grands défis de la biologie puisque le logiciel a décrit la structure tridimensionnelle des protéines avec un taux de succès médian de 92% pour l’ensemble des protéines soumises et de 87% pour les protéines dont la structure était jugée comme étant particulièrement difficile à résoudre ! Est-ce le début d’une nouvelle ère ? C’est ce qu’affirment en tout cas certaines personnes qui travaillent dans le domaine. (suite…)
Le 30 novembre dernier, les résultats d’une compétition de prédiction de la structure des protéines appelée « CASP » (Critical assessment of structure prediction) ont été rendus publics. À chaque deux ans, une centaine d’équipe dans le monde tentent de découvrir à l’aide de logiciels la structure tridimensionnelle de certaines protéines que l’on a réussi à identifier par d’autres moyens expérimentaux. Depuis 2006, les taux de succès de ces logiciels oscillaient entre 30 et 40%. Puis, en 2018, le logiciel AlphaFold de la branche Deep Mind de Google, qui utilisent les réseaux de neurones artificiels (le fameux « deep learning »), a fait une entrée fracassante dans la partie avec un taux de succès de près de 60% ! C’est la même approche que Deep Mind avait déployé dans son logiciel AlphGo qui avait battu en 2016 le champion du monde du jeu de Go. Or les performances d’AlphaFold(2), la version 2020 du logiciel, lors de la compétition de cette année marquent, selon certains, un tournant majeur dans l’un des plus grands défis de la biologie puisque le logiciel a décrit la structure tridimensionnelle des protéines avec un taux de succès médian de 92% pour l’ensemble des protéines soumises et de 87% pour les protéines dont la structure était jugée comme étant particulièrement difficile à résoudre ! Est-ce le début d’une nouvelle ère ? C’est ce qu’affirment en tout cas certaines personnes qui travaillent dans le domaine. (suite…)
Du simple au complexe | Comments Closed